科研进展
Genome Biology丨郝沛课题组合作建立深度学习的神经网络RNA编辑识别模型
发表日期: 2023-04-20
4月17日,研究所郝沛课题组和中国科学院分子植物科学卓越创新中心李轩课题组合作,在国际学术期刊Genome Biology发表了题为DeepEdit: single-molecule detection and phasing of A-to-I RNA editing events using nanopore direct RNA sequencing的研究论文。该研究利用深度学习神经网络方法,训练构建了转录组RNA的A>I 编辑识别的深度学习算法:DeepEdit。这项成果是国际上首次直接对转录组RNA测序数据进行单分子水平的A>I编辑检测及对编辑事件的相位分析,为转录组直接测序鉴定RNA编辑和研究RNA编辑功能提供了全新的技术方法。

A>I 的RNA编辑是动物中最常见的RNA修饰方式,在转录后层面调控RNA的结构和功能。在编辑过程中,腺嘌呤被脱氨酶作用转化为次黄嘌呤。由于次黄嘌呤在翻译过程中被识别为鸟嘌呤,因此A>I的RNA编辑可以导致蛋白质序列的变化。当前,通过二代测序的短读测序分析cDNA(由RNA逆转录获得)的单核苷酸变异是检测RNA的 A>I修饰的主要方法。然而,这种方法存在诸多不足,包括无法确定编辑碱基的相位信息、短读长度导致的假阳性编辑位点以及计算复杂度高、难以解释RNA编辑与其他转录后调控事件(如可变剪接)之间的关系。
本研究针对RNA最常见A>I的RNA编辑,利用深度学习神经网络方法,建立转录组 A>I编辑的识别定量模型。研究人员利用裂殖酵母材料,包括野生型和转化人源ADAR2酶的实验型,通过二代测序筛选高置信度的A>I编辑位点,比较位点上的高丰度A>I事件(实验型)和无修饰(野生型)的信号特征。基于特异性碱基错配模型对样本进行标注,分离提取编辑碱基与之对应的信号特征。使用深度学习神经网络方法进行训练,建立了识别RNA A>I编辑修饰结构的信号识别模型和分析工具(DeepEdit)。
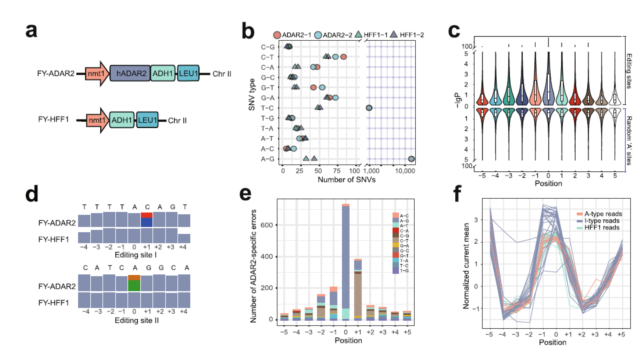
图例:A>I编辑对纳米孔测序读段特征的影响
DeepEdit是一种基于神经网络的算法模型,可以在单分子水平上检测和分析RNA编辑事件,并解决转录本上RNA编辑事件的相位问题。它可以识别A>I编辑事件,并且具有高度的准确性和鲁棒性。DeepEdit模型可以应用于酵母和人类等多物种的纳米孔RNA测序数据,有望在RNA研究领域中得到广泛应用,促进我们对影响人类健康的疾病相关发生机制的研究。
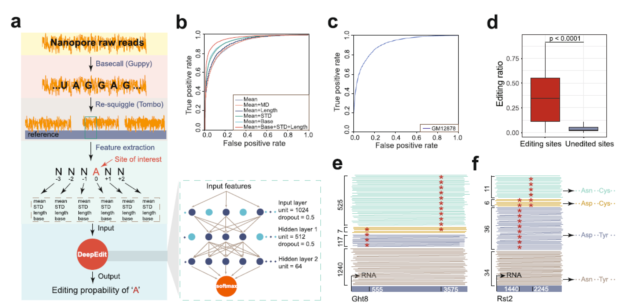
图例:DeepEdit在单个Nanopore测序读段中识别A>I RNA编辑事件
中国科学院分子植物科学卓越创新中心陈龙现、荆新云,研究所欧亮,西奈山伊坎医学院孔艺萌为本文的共同第一作者。研究所郝沛研究员和中国科学院分子植物科学卓越创新中心李轩研究员为共同通讯作者。该研究工作获得了国家重点研发计划、国家自然科学基金和中科院先导项目的支持。
论文链接:
https://genomebiology.biomedcentral.com/articles/10.1186/s13059-023-02921-0
附件下载: